GO enrichment Test
仅供参考。 GO enrichment检验方法一般是超几何分布检验,虽然原理简单但是经常发现不同软件的结果差别很大。这次对常用的三个软件,clusterProfiler,DAVID和BINGO,简单测试了一下GO enrichment在Cellular Component类别上的结果差别。
随机生成240的human gene entrez ID, 文件 geneList.txt。
P值多重检验校正方法,FDR(BH)adjustment。
1 clusterProfiler, R bioconductor package。
library("clusterProfiler")
entrez_ID<-read.table("GeneList.txt",header=T,stringsAsFactors=F)[,1]
enrich_GOs<-enrichGO(gene=entrez_ID,organism="human",ont="CC",pvalueCutoff=0.05,readable=TRUE)
clusterProfiler_output=enrich_GOs@result
结果得到5个显著富集的GO term,具体信息省略。
GOTERM geneCount
GO:0097381 5
GO:0001750 6
GO:0045121 11
GO:0098857 11
2 使用David GOTERM_CC_ALL (adjustment method FDR)
FDR校正后,只有两个显著Term,放宽条件为Benjamini adjustment,多了两个term。
GOTERM geneCount
GO:0043226 169
GO:0043227 159
GO:0097381 5 (Benjamini)
GO:0001750 7 (Benjamini)
3 使用cytoscape 的plugin, BINGO
7个显著Term,但是GO term内注释到的基因数目比以上两个软件得到的较少,估计是GO database的问题。
GOTERM geneCount
GO:0044424 148
GO:0005622 149
GO:0022627 4
GO:0070013 33
GO:0005654 22
GO:0031974 34
GO:0044451 16
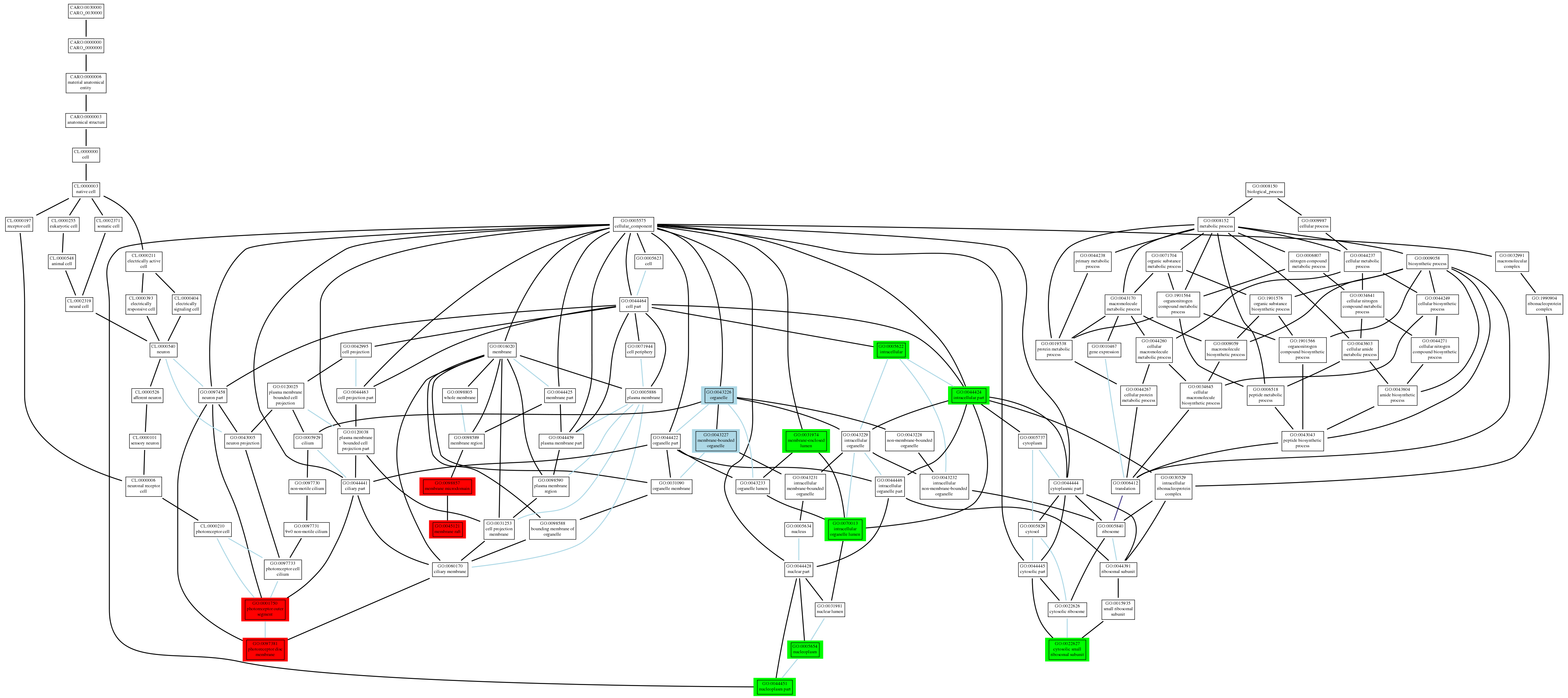
比较三个软件得到的 overrepresented Terms之间的关系
红色,clusterProfiler显著Terms。浅蓝,David显著Terms。绿色,BINGO显著Terms。
library("RamiGO")
goIDs <- c("GO:0097381","GO:0001750","GO:0045121","GO:0098857",
"GO:0043226","GO:0043227","GO:0044424","GO:0005622","GO:0022627",
"GO:0070013","GO:0005654","GO:0031974","GO:0044451")
color <- c(rep("red",4),rep("blue",2),rep("green",7))
pngRes <- getAmigoTree(goIDs=goIDs, color=color, filename="example", picType="png", saveResult=TRUE)
clusterProfiler富集的是direct terms。background用的是org.Hs.eg.db, 一年两次更新。
David的结果,选的GOTERM_CC_ALL的结果且用FDR adjustment,因为direct terms中没有显著的富集GOTERMs。用DAVID做GO enrichment时候还是适合选Benjamini的adjustment,Benjamini adjustment显著的GO terms有两个与clusterProfiler一致,GO:0097381和GO:0001750。