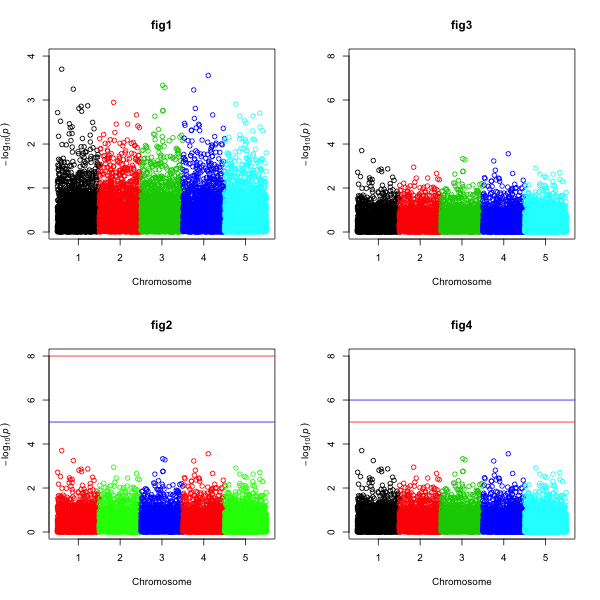
以R作GWAS的manhattan图
来自gettinggeneticsdone(blogspot)上的一段ManhattanPlot的R代码,稍加修改了一下以方便自己的使用。blogspot被墙了,所以没去找原作者的Rscript地址。</br>
我弄得修改版在这里。
https://github.com/geneticswithme/hello_genetics/blob/master/manhattan_z.R
加载上manhattan这个函数后,就可以直接使用这个函数对PLINK关联分析结果文件作manhattan图。
这里我随机生成一个assoc数据框,以解释manhattan这个函数的使用。</br>
manhattan这个函数的输入必须为dataframe,且包含“CHR”,“SNP”,“BP”和“P”这四个header。
setwd("G:/manhattan/test/")
source("manhattan_z.R")
#生成测试用的dataframe
set.seed(20)
df<-data.frame("CHR"=rep(1:5,c(2000,2000,2000,2000,2000)),
"SNP"=paste("SNP",1:10000,sep=""),
"BP"=rep(1:2000,5),
"P"=runif(10000,0,1))
png("manhattan.png",width=500,height=500)
par(mfcol=c(2,2))
manhattan(df,col=c(1:5),main="fig1") #fig1
manhattan(df,col=c("red","green","blue"),ymax=8,main="fig2") #fig2
manhattan(df,col=c(1:5),ymax=8,suggestiveline=F,genomewideline=F,main="fig3") #fig3
manhattan(df,col=c(1:5),ymax=8,suggestiveline=6,genomewideline=5,main="fig4") #fig4
dev.off()#哈哈~~没有显著位点(演示数据而已~)