利用PLINK进行GWAS分析(一)
仅供参考。
利用PLINK软件完成整个GWAS分析,其实一点也不复杂。只要按照既定的分析流程,都可以得到分析结果。但是为了使自己的工作真正有意义,必须要理解每一步处理的原理和目的, 从而根据不同的试验设计,调整数据处理过程。
这里列出的GWAS分析流程以Illumina 的SNP数据为例。如果你手头上的是Affy或者其他平台的芯片数据,处理方法也是类似。 1、原始数据整理为PLINK可识别的文件 2、利用PLINK读取整理后的数据 3、利用PLINK进行数据质控 4、利用PLINK计算群体结构 5、利用PLINK进行关联分析 6、对分析结果进行图形展示
首先进行数据整理。需要将Illumina的原始数据整理为可被PLINK软件识别的PED/MAP文件类型。PED文件和MAP文件都是文本文件,可以被文本编辑器打开。PED文件主要包括每个试验个体的SNP数据和试验群体的家系等信息。MAP文件主要列出的是SNP位点的名称和位置信息。 关于PED/MAP文件的详细格式,PLINK软件的document中有详细说明,这里不再叙述。
这里的数据处理都是在Windows系统下进行。但是还是建议大家学习linux,渐渐把自己的工作移到linux平台下。
1、MAP文件的获得
原始数据中,一般都会有MAP文件,但是往往参考基因组版本老旧。所以需要对MAP文件进行更新。 有个网站SNPchiMp,提供几种农业动物的SNP芯片数据的MAP文件下载。根据网站提示可以很容易获得最新版本的SNP物理位置数据,稍微编辑一下即可得到MAP文件。
SNPchiMp网页的截图
2、下载GenomeStudio软件
GenomeStudio软件是用来读取Illumina 芯片原始数据,进行数据查看的,输出数据的软件。可以在Illumina官网下载安装。因为分析的是SNP芯片数据,所以只需注册GenomeStudio的Genotyping module即可。
GenomeStudio的说明详见GenomeStudio,Genotyping Module的User Guide。 在Genotyping Module上安装PLINK插件,可以由此将原始数据输出为PLINK的PED格式的SNP数据。
3、利用GenomeStudio输出PED文件
Analysis→Reports→Repost Wizard
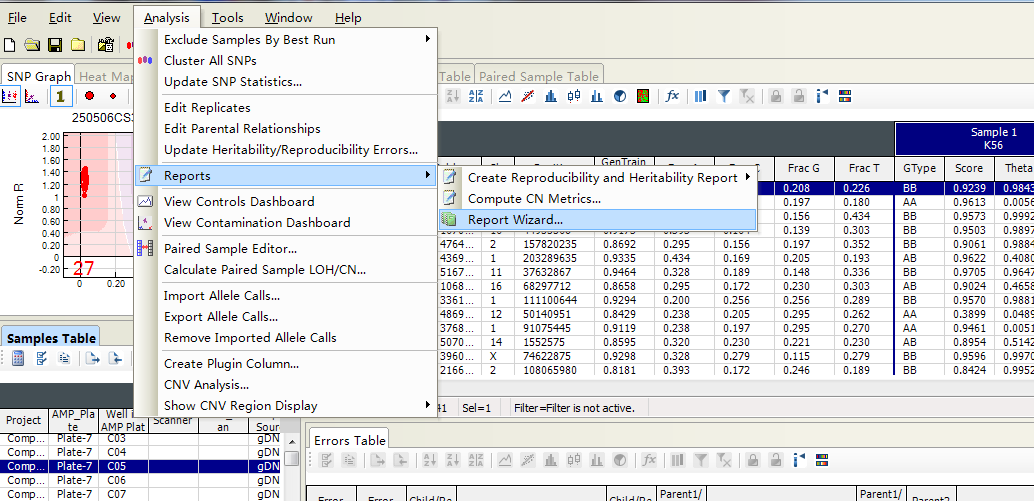
自定义输出格式,Custom Report→PLINK Input Report
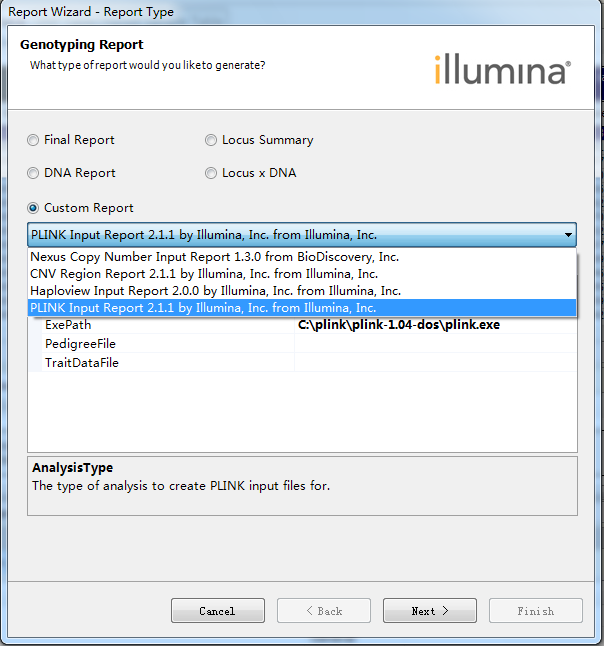
按照提示操作输出PED文件。
4、整理MAP文件和PED文件
最好将MAP文件和PED文件统一命名,例如test.ped和test.map。注意,文件格式一定要符合PLINK软件的要求。
如需查看MAP和PED文件,可以下载Notepad++软件,利用Notepad++软件打开。Windows自带的文本编辑器不容易打开比较大的文本文档。
5、PLINK软件的下载与安装
下载地址PLINK v1.07,这里下载的是1.07版本,目前最新的PLINK版本为1.09。这两个版本之间做了很大的改变,1.09版本明显好用了很多。但读取数据和基本分析命令都是差不多的,这里仅以1.07版本举例说明。根据自己的操作系统下载相应版本的软件,Windows系统的,下载MS-DOS版本。
下载好的PLINK软件包加压后得到的文件夹包括plink.exe和gPLINK.jar两个程序,gPLINK.jar是依赖Java的图形界面化程序,plink.exe是在dos下运行的命令行程序。两者本质一样,运行方式不同。个人觉得plink.exe更灵活好用一些。
6、运行plink.exe
windows系统下同时按下键盘上的【开始】+R两个键,打开运行,输入cmd,回车后进入命令提示符界面。

整理好得文件为test.ped和test.map文件。放入G盘下的test文件夹中,plink.exe程序同样放入该文件夹中。
命令提示符后输入G:进入G盘目录下,输入cd test进入test目录,输入dir,查看当前目录的情况,可以看到有三个文件,分别是test.ped,test.map和plink.exe。
这个时候直接输入plink,可以见到如下情况,提示没有输入文件。
 输入
输入plink --file test,就可以看到plink读入的test.ped和test.map两个文件的结果。